Домашнее задание 2¶
Выполнил: Ким Адамейко, группа мАДБМ16
import time
import __main__
import os
import pymol
__main__.pymol_argv = [ 'pymol', '-cp' ]
pymol.finish_launching()
from pymol import cmd
from IPython.display import Image, display, HTML
from __future__ import print_function
def prepareImage(filename='tmp.png', width=500, height=500, sleep=0.5, show=False):
filename = filename[0] if isinstance(filename, tuple) else filename
if filename[-3:] != 'png': filename += '.png'
cmd.viewport(width, height)
cmd.ray(width, height)
cmd.png(filename)
time.sleep(sleep)
if show:
display(HTML('<img src="%s?v=%d"/>' % (filename, int(time.time()))))
def img_row(img_data): # tuples of (filename, title)
img_files, titles = zip(*img_data)
display(HTML("<table>" +
('<tr>' + ''.join(['<th style="text-align: center;">' + title + '</th>'
for title in titles]) + '</tr>' if titles else '') +
'<tr>' + ''.join(['<td><img src="' + img + '.png?v=' + str(int(time.time())) + '"/></td>'
for img in img_files])+'</tr></table>'))
Часть 2. Поиск водородных связей¶
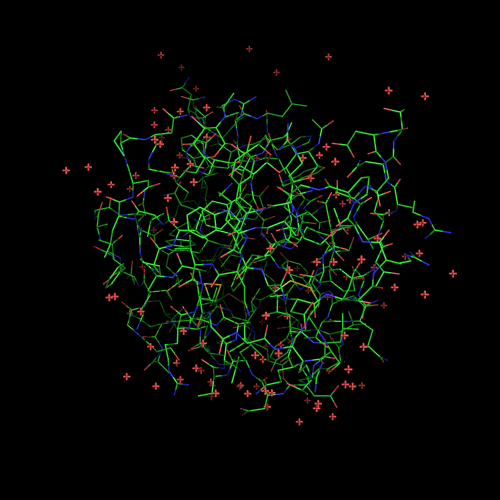
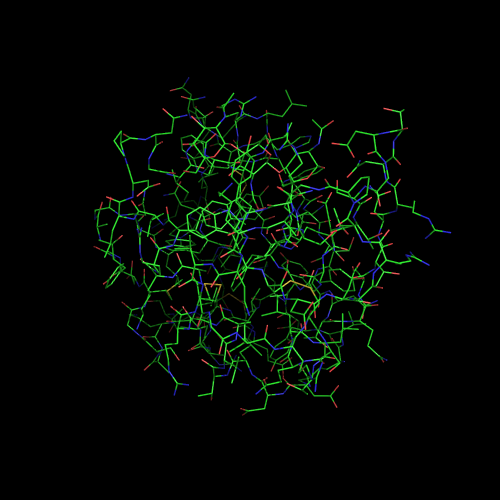
Нужно проанализировать связи между хитоолигосахаридами и лизоцимом радужной форели.
images = (('1LMP_01', 'Полная картина'), ('1LMP_02', 'Без несвязанных атомов'))
cmd.reinitialize()
cmd.fetch("1LMP", async=0) # загружаем файл из интернета по идентификатору PDB в синхр.режиме
prepareImage(images[0])
cmd.hide('nonbonded') # отключаем отображение несвязанных атомов (ионов, воды)
prepareImage(images[1])
img_row(images)
С помощью сайта RCSB PDB [2] можно изучить связи между лигандами и белком, пройдя по ссылке Ligand Explorer и воспользовавшись соответствующим Java Web Start приложением. В этом приложении также можно получить карту контактов
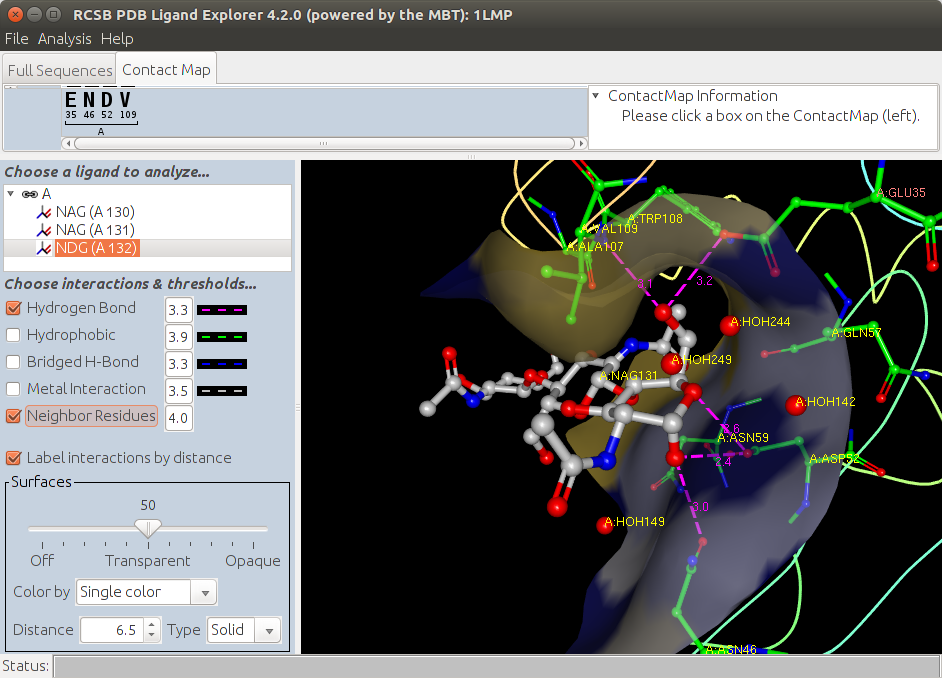
Попробуем получить подобные картинки с помощью PyMol
cmd.reinitialize()
cmd.fetch("1LMP", async=0)
cmd.remove('solvent') # убираем атомы воды
cmd.h_add('all') # добавим водороды
cmd.extract('ligands', 'het') # экстрагируем атомы лиганда (т.н. гетероатомы) в объект ligands
cmd.orient('1lmp') # ориентируем вдоль "главных осей" белка
cmd.turn('y', 200) # поворот (красивый угол подобрал на локальной машине)
cmd.turn('x', 30) # поворот
cmd.show_as('sticks', 'ligands') # лиганд изобразим толстыми палочками
cmd.show_as('surface', '1lmp') # белок изобразим в виде полупрозрачной поверхности
cmd.set('transparency', 0.5)
prepareImage('1LMP_03', 1000, 500, show=True)
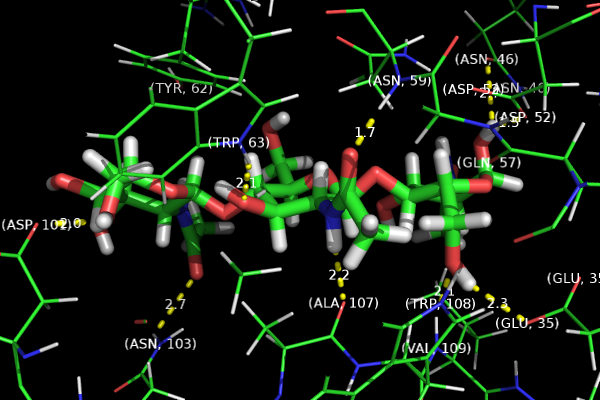
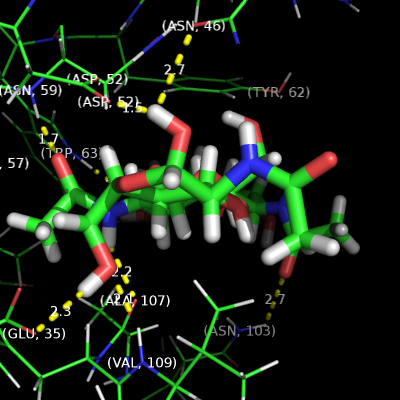
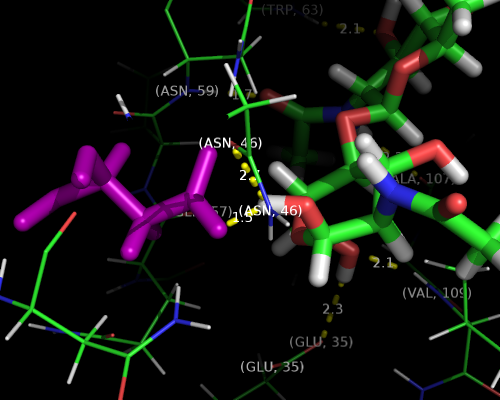
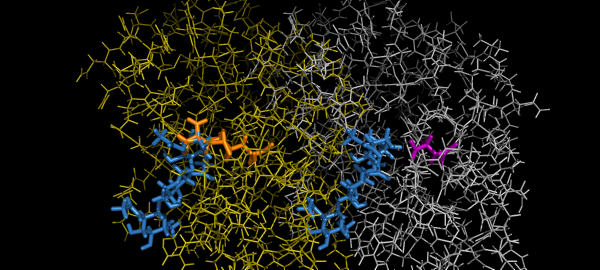
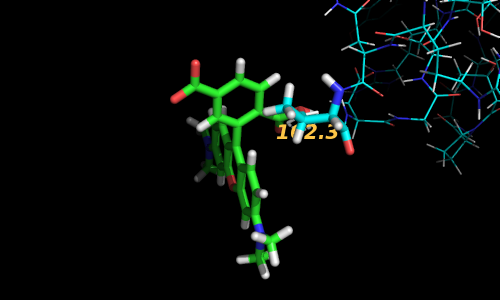
Приблизим участок взаимодействия, изобразим:
- остатки в пределах 4 Å от лиганда
- водородные связи (по [3]) между электроотрицательными атомами белка и лигандом с помощью функции distance()
- c максимальной длиной связи 3.2 Å (общепринятый порог, который, однако, не гарантирует правильность всех построенных связей)
- и режимом 2.
- подписи к взаимодействующим атомам белка, которые позволят идентифицировать остатки
Посмотрим на лиганд вдоль двух "осей" сайта связывания
cmd.set('transparency', 1)
cmd.select('don', '(elem n,o and (neighbor hydro))') # доноры
cmd.select('acc', '(elem o or ((elem n) and (not (neighbor hydro))))') # акцепторы
cmd.distance('HBA', '(ligands and acc)', '(1lmp and don)', 3.2, 2) # связи, где акцепторы в лиганде
cmd.distance('HBD', '(ligands and don)', '(1lmp and acc)', 3.2, 2) # связи, где акцепторы в белке
cmd.set('label_color', 'white', 'HBA') # цвет и размер шрифта подписей на связях
cmd.set('label_color', 'white', 'HBD')
cmd.set('label_size', 16)
cmd.reset() # сброс ориентации камеры
cmd.show('lines', 'byres(1lmp w. 4 of ligands)') # покажем близлежащие к лиганду атомы белка тонкими линиями
cmd.set('label_shadow_mode', 0) # чтобы подписи не отбрасывали теней
cmd.select('contact', '(((acc or don) within 3.5 of ligands) and (not ligands))') # атомы белка, конт.через вод/св
cmd.do('label contact, "(%s, %s)" % (resn, resi)') # пометим их (назв, номер остатка)
cmd.orient('ligands') # сориентируем вдоль главной "оси" лиганда
cmd.turn('x', -90) # доп. повороты (подбирались на локальной машине)
cmd.zoom('ligands')
prepareImage('1LMP_04', 600, 400)
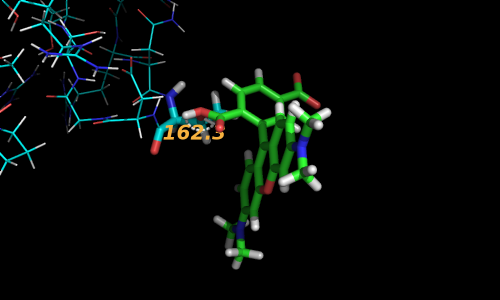
cmd.turn('y', -100) # вид сбоку
cmd.zoom('ligands')
prepareImage('1LMP_05', 400, 400)
img_row([('1LMP_04','Вид вдоль главной оси лиганда'), ('1LMP_05','Вид сбоку')])
Часть 3. Мутагенез¶
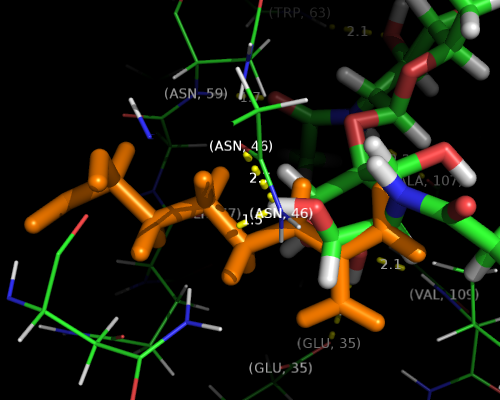
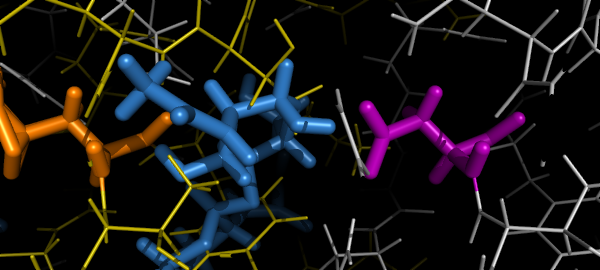
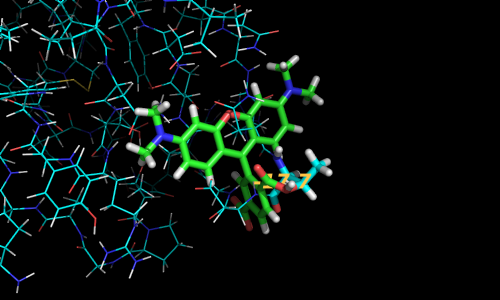
Рассмотрим, например, аспаргинин в позиции 52, он находится в близком контакте с лигандом (расстояние ~1.5 Å). Мутируем этот остаток так, чтобы он занимал больше пространства, и т.к. в этой части "кармашка" белка лиганду разворачиваться или двигаться особенно негде, можно предположить, что связь будет потеряна. Можно взять, например, аргинин, однако заметим, что для такой мутации остатка замена должна произойти в двух нуклеотидах.
images = [('mut_before', 'До мутации'), ('mut_after', 'После мутации')]
mut_resi = 52 # выбор исходного остатка по номеру
mutable_where_to = 'ARG' # выбор целевого остатка по названию
cmd.select('mut', 'resi %d or ligands within 5 of resi %d' % (mut_resi, mut_resi)) # выбор области для зума
cmd.zoom('mut')
cmd.orient('mut')
cmd.turn('x', 90)
cmd.colour('purple','resi %d' % mut_resi ) # окрашиваем и утолщаем мутируемый остаток,
cmd.show_as('sticks','resi %d' % mut_resi ) # чтобы сделать его заметнее
prepareImage(images[0], 500,400)
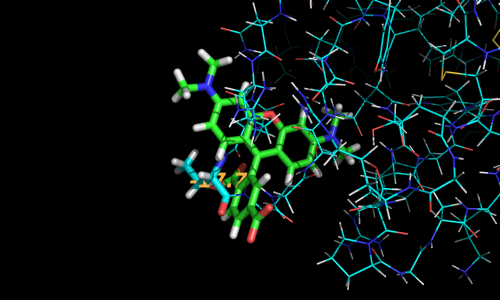
cmd.wizard("mutagenesis") # основной код процедуры мутации
cmd.do("refresh_wizard")
cmd.do('cmd.get_wizard().do_select("resi %d")' % mut_resi) # выбор мутируемого остатка
cmd.do('cmd.get_wizard().set_mode("%s")' % mutable_where_to)# выбор целевого остатка
cmd.do("refresh_wizard")
cmd.do("cmd.get_wizard().apply()") # применение мутации
cmd.do("cmd.set_wizard()")
cmd.colour('orange','resi %d' % mut_resi ) # снова окрашиваем
cmd.move('x', -3) # компенсируем сдвиг камеры (причину не выяснил)
prepareImage(images[1], 500, 400)
img_row(images)
cmd.save( "./1lmp_mut.pdb", 'all')
Часть 4. Анимация¶
Для начала скачаем утилиту ffmpeg для создания видео из картинок
%%bash
if ! hash ffmpeg 2>/dev/null; then
mkdir -p '../bin/ffmpeg/'
if [ ! -f '../bin/ffmpeg/ffmpeg.tar.xz' ] ; then
echo 'loading...'
wget -nv -O '../bin/ffmpeg/ffmpeg.tar.xz' https://johnvansickle.com/ffmpeg/releases/ffmpeg-release-32bit-static.tar.xz
fi
echo 'extracting...'
tar xJf '../bin/ffmpeg/ffmpeg.tar.xz' -C '../bin/ffmpeg/' --strip-components 1
rm '../bin/ffmpeg/ffmpeg.tar.xz'
echo 'done.'
fi
chmod 755 -R '../bin/ffmpeg/'
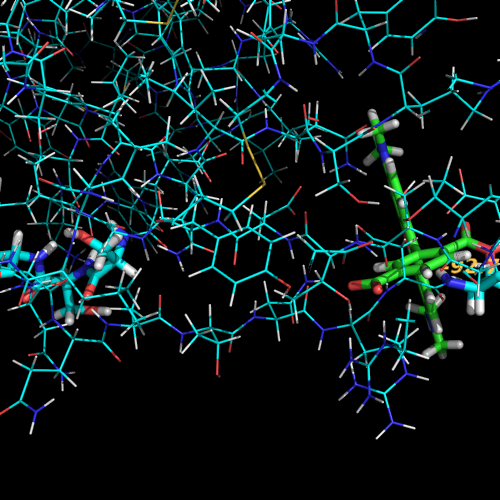
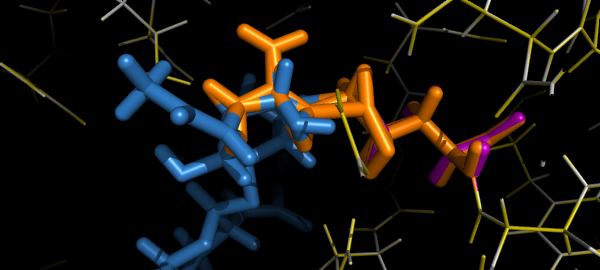
- Загрузим оригинальный и мутированный белок
- Подготовим объекты выделения для удобной манипуляции
- Раскрасим белки и лиганды, лиганды и место мутации покажем толстыми палочками
cmd.reinitialize()
mut_resi = 52 # выбор исходного остатка по номеру
cmd.fetch('1lmp', 'ori')
cmd.load('./1lmp_mut.pdb', 'mut')
cmd.remove('solvent') # убираем атомы воды
cmd.h_add('all') # добавим водороды
cmd.select('lig1', 'ori and het')
cmd.select('lig2', 'mut and het')
cmd.select('res', 'resi %d' % mut_resi)
cmd.select('mutspot', 'res + (lig* within 5 of res)')
cmd.hide('everything')
cmd.show('lines', '(mut | ori) & !lig*')
cmd.show('sticks', 'lig* | res')
cmd.colour('olive', 'mut')
cmd.colour('silver', 'ori')
cmd.colour('skyblue', 'lig*')
cmd.colour('purple', 'ori & res')
cmd.colour('orange', 'mut & res')
cmd.set('transparency', 0.7)
- Подготовим стартовый кадр. Пока ещё не снимаем фильм, можно использовать
translate()без именованного параметра object!
width, height = 600, 300
cmd.orient('mutspot')
cmd.turn('x', 60)
cmd.turn('y', 10)
cmd.translate('[-50, 0, 0]', 'mut')
cmd.zoom('*', -14)
cmd.move('x',-4)
prepareImage('tmp', width, height, show=True)
- Дальнейшая последовательность действий сначала долго оттачивалась на локальном компьютере. Некоторый опыт...
- Не все команды возможно корректно исполнить вне простой обёртки
cmd.do(), сначала я пытался, потом сдался. Особенно это касается именованного параметраobject, который не может быть заменён позиционным вторым параметром, отвечающим за выделение -- это не сработает при съёмке фильма! - Функции
util.mrock()иutil.mroll()я не смог заставить выдать результат, который мне бы понравился, поэтому вращал сцену вручную. - Иногда, если переходить к новому кадру слишком быстро, пока Pymol не успел интерполировать предыдущие, в этом новом кадре сцена может быть выставлена некорректно. Решил проблему отключением авто-интерполяции.
- Не все команды возможно корректно исполнить вне простой обёртки
n_frames = 360
cmd.set('matrix_mode', 1)
cmd.set('ray_trace_frames', '0')
cmd.set('movie_auto_interpolate', '0')
cmd.viewport(width, height)
cmd.mset('1 x%d' % n_frames)
cmd.frame('1')
cmd.mview('store')
cmd.do('mview store, object=mut')
cmd.frame('%d' % (n_frames // 5))
cmd.do('translate [30, 0, 0], object=mut')
cmd.center('mutspot')
cmd.zoom('*', -10)
cmd.mview('store')
cmd.do('mview store, object=mut')
cmd.frame('%d' % (n_frames // 2))
cmd.do('translate [20, 0, 0], object=mut')
cmd.do('mview store, object=mut')
cmd.zoom('mutspot', -2)
cmd.mview('store')
cmd.frame('%d' % (n_frames))
cmd.do('mview store, object=mut')
cmd.zoom('mutspot', -2)
cmd.center('mutspot')
cmd.mview('store')
cmd.frame(n_frames//3)
cmd.center('mutspot')
cmd.zoom('mutspot', -2)
cmd.mview('store')
for i in range(n_frames//2+1, n_frames+1, 2):
cmd.frame(i)
cmd.do('turn y, -8')
cmd.mview('store')
cmd.do('mview interpolate')
cmd.do('mview interpolate, object=mut')
- Сохраним кадры и выведем несколько ключевых, чтобы проверить, что всё хорошо
! mkdir -p ./frames/
! rm ./frames/*
cmd.set('ray_trace_frames', '1')
cmd.set('antialias', '1')
cmd.mpng('./frames/1lmp_frame')
images = []
for i in [7, 5, 3, 2]:
frame_name = '1lmp_frame%04d' % (n_frames // i)
! cp './frames/{frame_name}.png' './{frame_name}.png'
images.append(('./' + frame_name, ''))
img_row(images)
- Наконец, собираем фильм
! '../bin/ffmpeg/ffmpeg' -y -i './frames/1lmp_frame%04d.png' -c:v libx264 -pix_fmt yuv420p -vf "scale=trunc(iw/2)*2:trunc(ih/2)*2" movie_1lmp.mp4
! rm ./frames/*
HTML('<video controls alt="PyMol Movie" src="movie_1lmp.mp4?" type="video/mp4">')
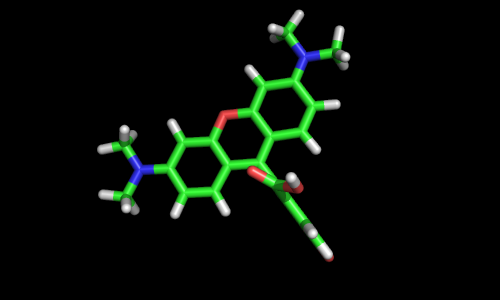
Часть 5. Метка TAMRA¶
Сложноэфирная связь: $\require{mhchem}\ce{R-COOH + HO-R' <=>[H+] R-COO-R' + H2O}$
Загрузим метку TAMRA, видим там карбоксильную группу ($\ce{-COOH}$)
! wget -nv -O './tamra.sdf' 'https://pubchem.ncbi.nlm.nih.gov/rest/pug/compound/cid/2762604/record/SDF/?response_type=save&record_type=3d'
cmd.reinitialize()
cmd.load('./tamra.sdf', 'tamra')
cmd.show('sticks')
cmd.zoom('*')
prepareImage('tamra_1', 500, 300)
cmd.turn('x', 60)
cmd.turn('y', 120)
prepareImage('tamra_2', 500, 300)
img_row([('tamra_1',''), ('tamra_2','')])
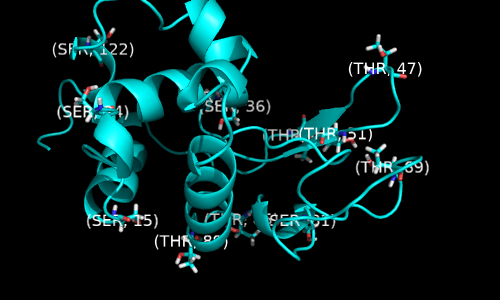
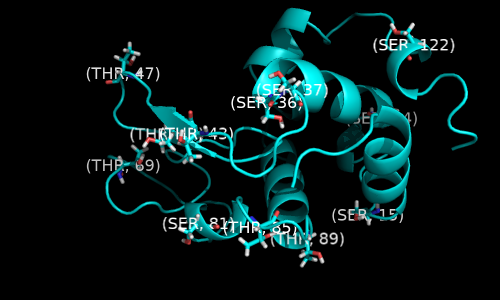
Затем загрузим снова наш белок, выделим все остатки серина и треонина (они содержат гидроксильную группу $\ce{-OH}$), находящиеся на поверхности белка. Нижеследующий скрипт, который отбирает такие остатки, взят с сайта PymolWiki [6]
%%writefile ./find_surface_residues.py
import pymol
from pymol import cmd, stored
import random
# https://pymolwiki.org/index.php/FindSurfaceResidues
def findSurfaceResidues(objSel="(all)", cutoff=2.5, doShow=False, verbose=False):
tmpObj="__tmp"
cmd.create( tmpObj, objSel + " and polymer");
if verbose!=False:
print("WARNING: I'm setting dot_solvent. You may not care for this.")
cmd.set("dot_solvent");
cmd.get_area(selection=tmpObj, load_b=1)
cmd.remove( tmpObj + " and b < " + str(cutoff) )
stored.tmp_dict = {}
cmd.iterate(tmpObj, "stored.tmp_dict[(chain,resv)]=1")
exposed = stored.tmp_dict.keys()
exposed.sort()
randstr = str(random.randint(0,10000))
selName = "exposed_atm_" + randstr
if verbose!=False:
print("Exposed residues are selected in: " + selName)
cmd.select(selName, objSel + " in " + tmpObj )
selNameRes = "exposed_res_" + randstr
cmd.select(selNameRes, "byres " + selName )
if doShow!=False:
cmd.show_as("spheres", objSel + " and poly")
cmd.color("white", objSel)
cmd.color("red", selName)
cmd.delete(tmpObj)
return exposed
cmd.extend("findSurfaceResidues", findSurfaceResidues)
cmd.disable('tamra')
cmd.set('label_size', 16)
cmd.set('label_shadow_mode', 0)
cmd.fetch('1lmp')
cmd.remove('solvent')
cmd.h_add('all')
cmd.hide('all')
cmd.select('prot', '!het')
cmd.do('run ./find_surface_residues.py')
cmd.do('findSurfaceResidues')
cmd.select('all_thr_ser', '(resn thr | resn ser) & exposed_res*')
cmd.do('label n. CA & all_thr_ser, "(%s, %s)" % (resn, resi)')
cmd.show('cartoon', 'all')
cmd.show('sticks', 'all_thr_ser')
cmd.orient('all_thr_ser')
cmd.zoom('all_thr_ser', -4)
cmd.deselect()
prepareImage('1LMP_06', 500, 300)
cmd.turn('y', 180)
prepareImage('1LMP_07', 500, 300)
img_row([('1LMP_06',''), ('1LMP_07','')])
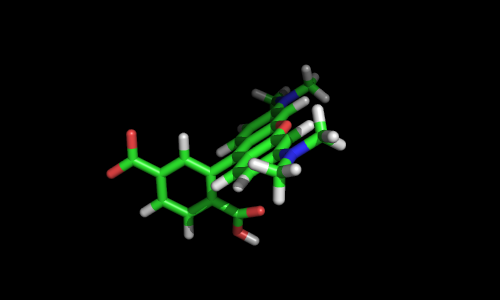
Выберем, например, треонин, остаток номер 47.
- Для совмещения выбираю кислород в гидроксильной группе треонина и углерод в карбоксильной группе TAMRA-метки.
- Сделав первые опыты, замечаю, что гидроксильная группа треонина не удаляется автоматически, что приводит к формированию странной связи. Удаляю её, выделяя соответствующие атомы в треонине.
- Для формирования связи TAMRA-метка копируется. Чтобы выделить атомы копии, можно обратиться к ним по имени
/1LMP///UNK/("unknown residue")
fusion_resi = '/1lmp//A/THR`47'
cmd.select("link_resi", "n. CB in %s" % (fusion_resi))
cmd.select("link_tamra", "n. O in tamra within 1 of n. H")
cmd.extract('hydroxyl', fusion_resi + '/H01 | ' + fusion_resi + '/OG1')
cmd.delete('hydroxyl')
cmd.fuse('link_tamra', 'link_resi')
cmd.select('fuse_tamra', '/1LMP///UNK/')
cmd.select('fuse_region', 'fuse_tamra | ' + fusion_resi)
cmd.deselect()
cmd.hide('all')
cmd.show('lines', 'prot & !fuse_region')
cmd.show('sticks', 'fuse_region')
cmd.turn('y', -20)
prepareImage('1LMP_08', 500, 300, show=True)
- Посмотрим издалека и увидим, что метка врезается в белок. К сожалению, готовой функции, детектирующей коллизии, я не нашёл, поэтому менял торсионный угол, "на глаз" подбирая оптимальное значение. В данном случае достаточно было повернуть на 180°
cmd.orient('fuse_region')
cmd.zoom('fuse_region', 2)
cmd.turn('y', 20)
prepareImage('1LMP_10', 500, 300)
cmd.turn('y', 180)
prepareImage('1LMP_11', 500, 300)
img_row([('1LMP_10',''), ('1LMP_11','')])
cmd.torsion(180)
cmd.orient('fuse_region')
cmd.zoom('fuse_region', 2)
cmd.turn('y', 120)
prepareImage('1LMP_12', 500, 300)
cmd.turn('y', 180)
prepareImage('1LMP_13', 500, 300)
img_row([('1LMP_12',''), ('1LMP_13','')])
Часть 6. Альфа-спираль¶
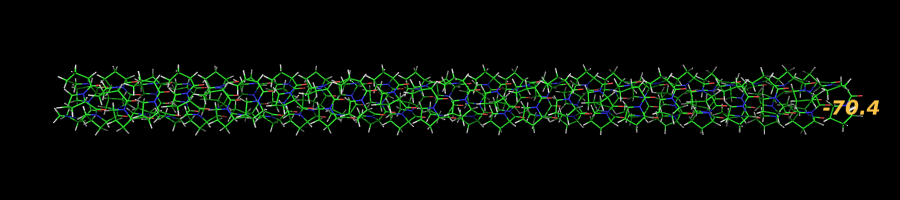
Я решил попробовать сделать поли-пролиновую альфа-спираль, поскольку пролин выделяется среди остальных аминокислот на картах Рамачандрана
cmd.reinitialize()
aa = 'pro' # пролин
cmd.fragment(aa)
# торсионные углы альфа-спирали
phi, psi = -60, -25
cmd.edit("i. %i & n. C" % 2)
cmd.edit("i. 2 & n. CA", "i. 2 & n. C")
n = 100 # число аминокислот в цепи
for i in range(2, n+1): # Присоединение аминокислот
cmd.edit("i. %i & n. C" % i)
cmd.do('editor.attach_amino_acid("pk1", "%s")' % aa)
time.sleep(0.1)
for i in range(2, n+1): # Установка торсионных углов
cmd.set_dihedral("i. %i & n. N" % i, "i. %i & n. CA" % i,
"i. %i & n. C" % i, "i. %i & n. N" % (i+1), phi)
cmd.set_dihedral("i. %i & n. C" % i, "i. %i & n. N" % (i+1),
"i. %i & n. CA" % (i+1), "i. %i & n. C" % (i+1), psi)
cmd.orient()
cmd.zoom('pro', -35)
prepareImage('poly_pro_01', 900, 200, show=True)
cmd.do('rotate z, 90, object=pro')
cmd.do('rotate x, 90, object=pro')
prepareImage('poly_pro_02', 500, 500, show=True)